CycloneSEQ-WT02纳米孔测序平台能够跨越细菌基因组中的复杂区域,能够实现一次性获得细菌基因组完成图。此外,CycloneSEQ技术在研究基因组重复区域、功能和适应性方面具有明显优势。

测试结果
PART.1
测序长度、质量结果
共测序三次,每次测序选取12个样本pooling在一张芯片。建库后用CycloneSEQ-WT02测序,随着测序时间的延长,数据产量逐步上升。平均每张芯片测序产生12Gb的数据量,平均每个样本产出1G左右数据量。
表1 三张CycloneSEQ芯片的测序结果
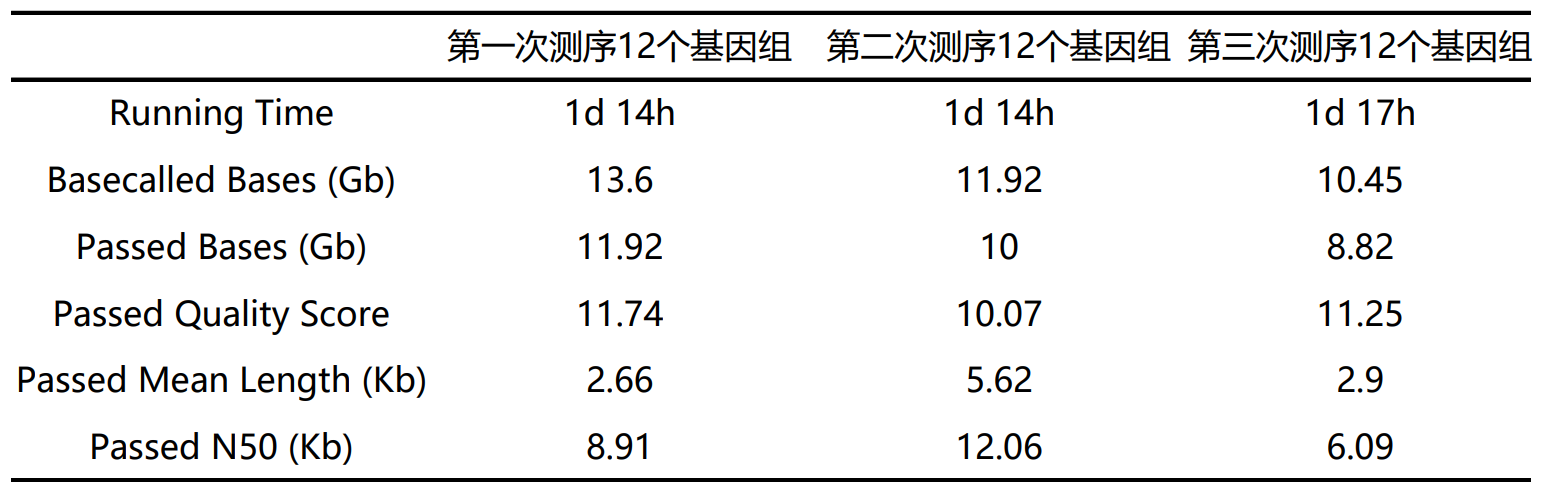
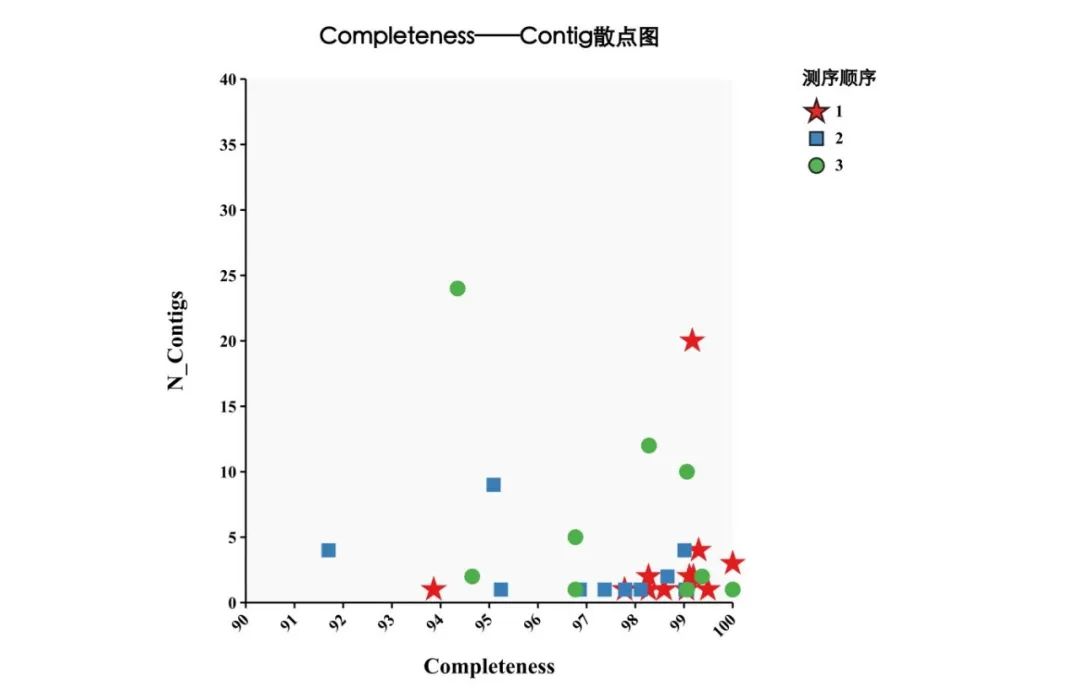
图1 36个基因组的完整度与基因组连续性(Contig数量)
测序产出数据随时间的变化有以下几个方面(以第一次测序产出为例):
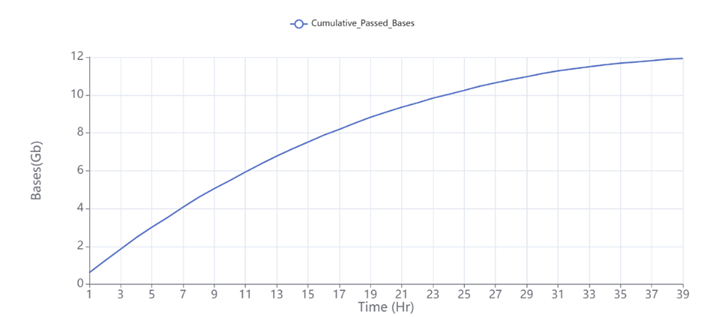
图2 第一次测序产出的base随时间增加曲线
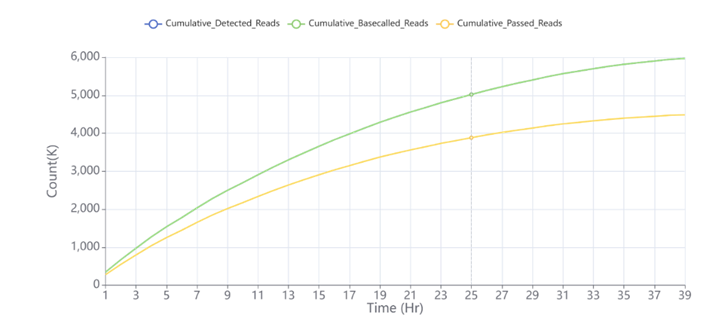
图3 第一次测序reads产出随时间增加曲线
表2 第一次测序每个 barcode产出信息
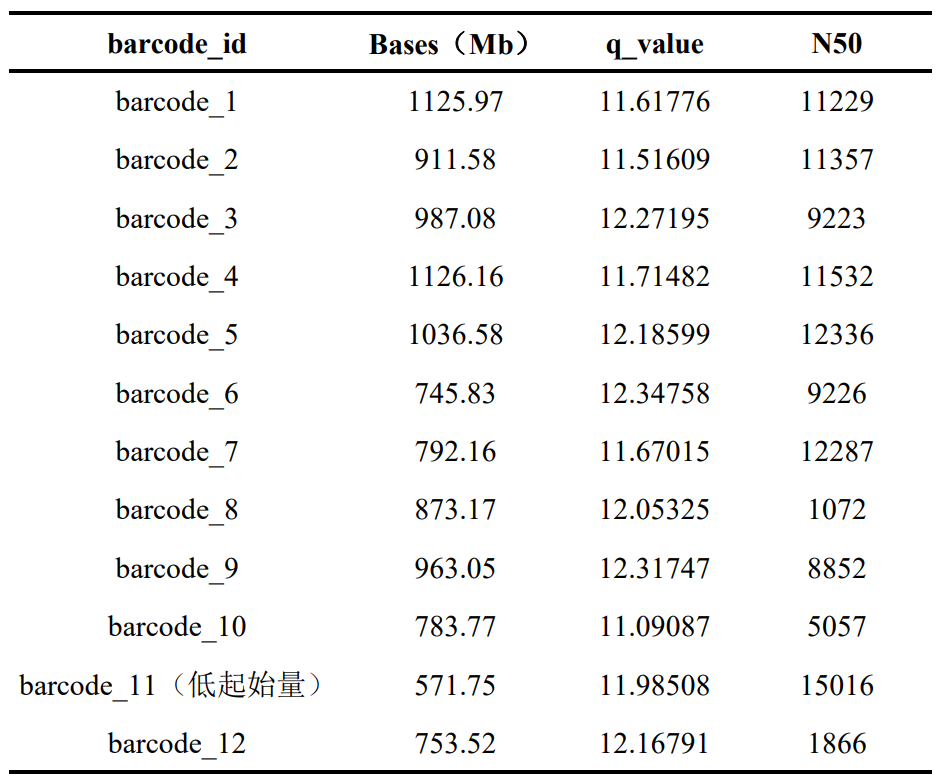
每次上样均含有1-3个低起始量样本,测序结果数据量与上样起始量基本对应。总体看来,单个样本产量较均一,读长比较稳定。
PART.2
仅CycloneSEQ测序基因组与参考基因组(完成基因组)比对
利用CycloneSEQ测序平台细菌基因组解决方案对某细菌(命名为Bacteria1,下同)测序分析得到了组装结果如表3、表4及图4、图5所示。
将Bacteria1菌株的CycloneSEQ测序组装基因组与该菌株的参考基因组做比对:
表3 Bacteria1组装策略和结果统计
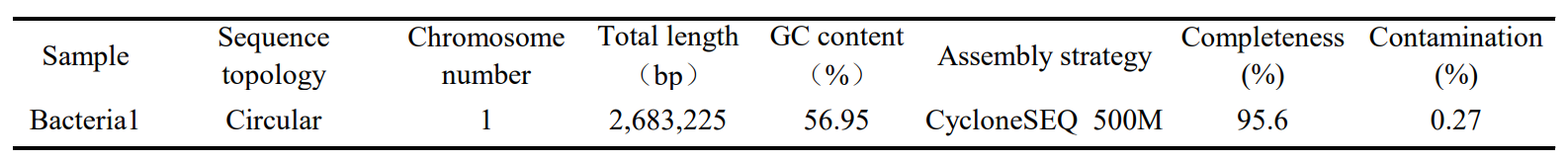
表4 Bacteria1与参考基因组比对统计
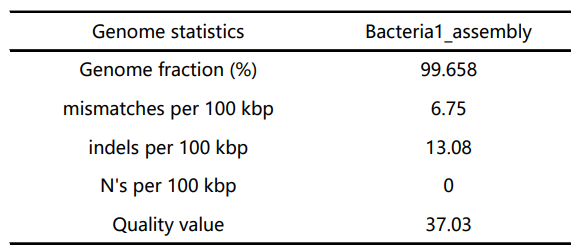
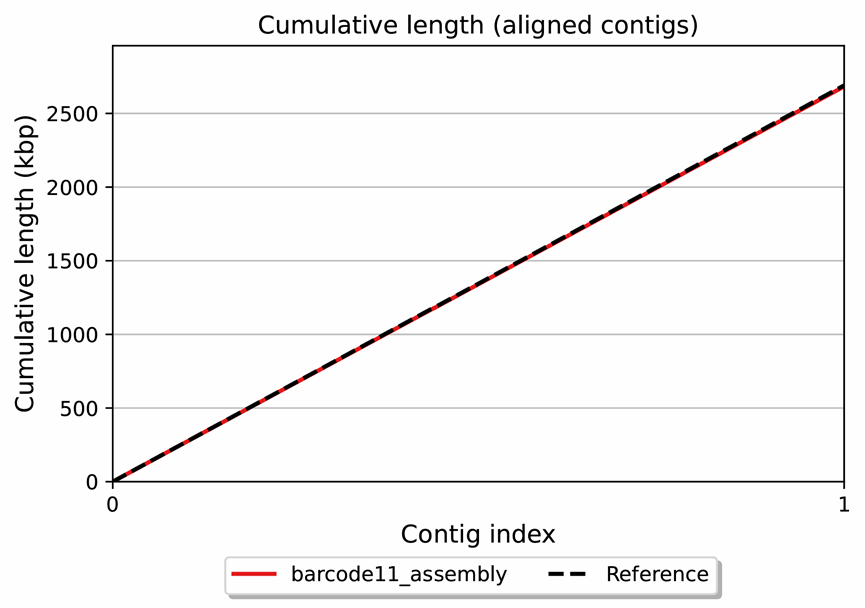
图4 Bacteria1与参考基因组比对情况
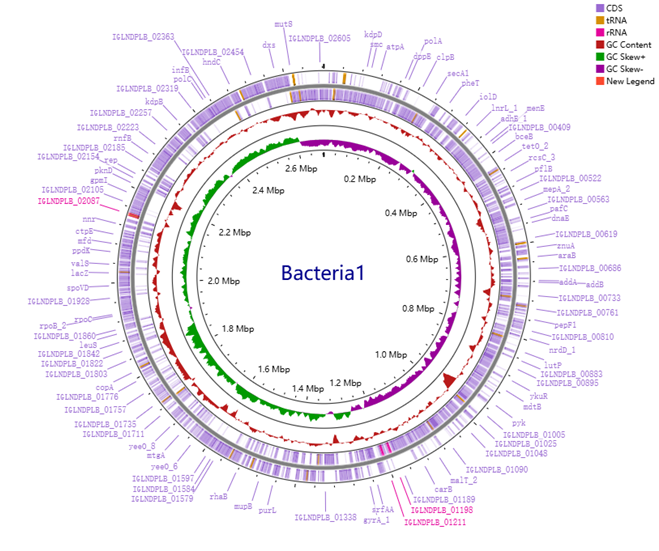
图5 Bacteria1基因组注释图谱
PART.3
环状质粒的完整测序及组装
Cyclone的长读长测序能够将菌株基因组外其他DNA序列准确并完整地测序。
表5 部分菌株质粒信息
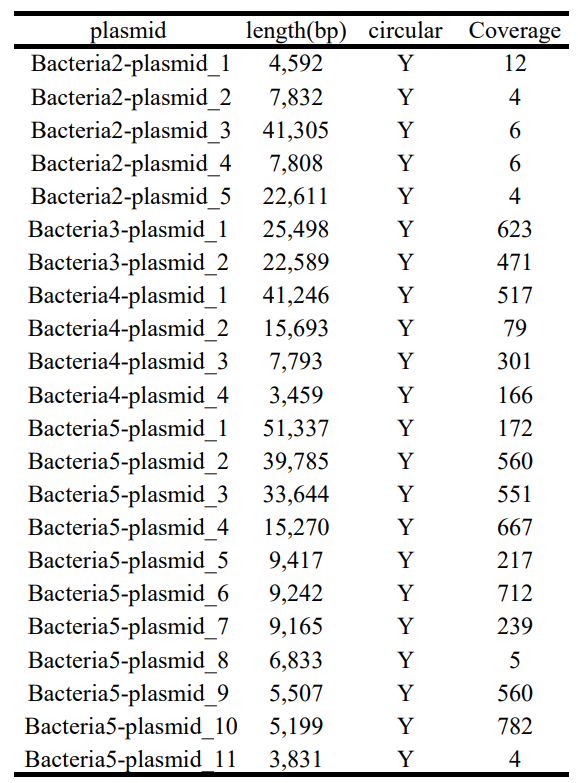
表6 Bacteria2菌株组装结果统计
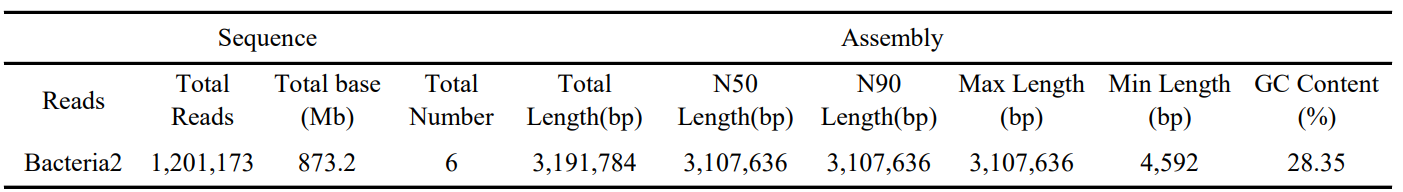
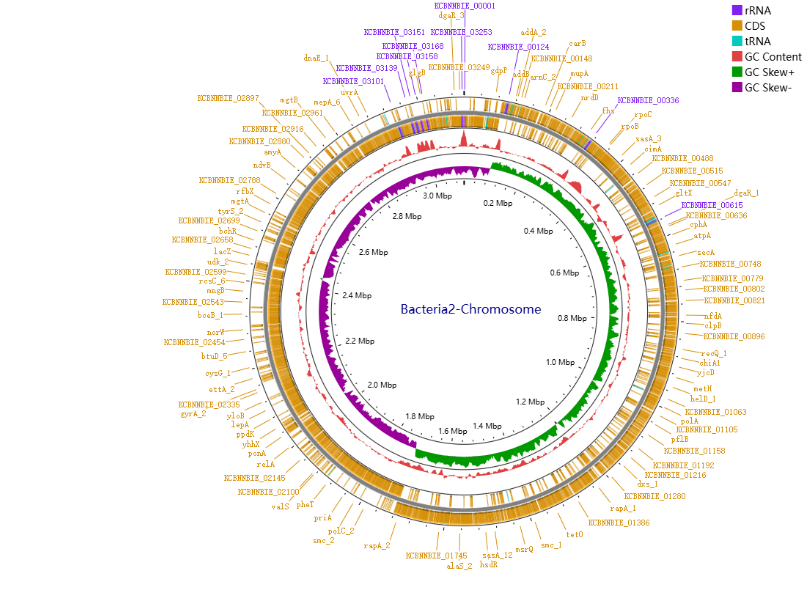
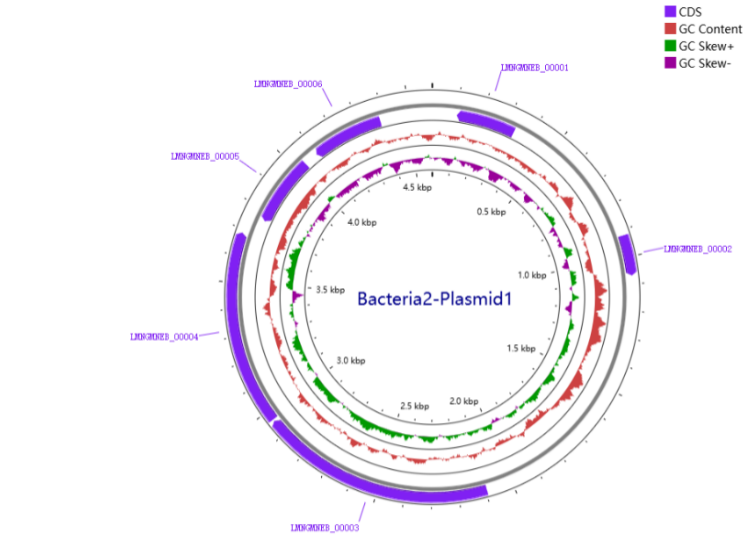
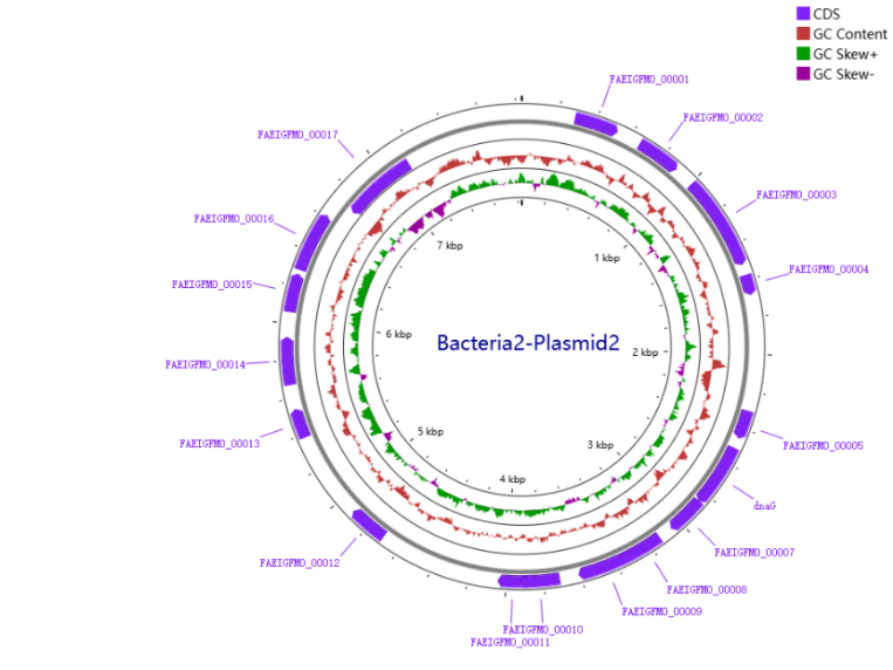
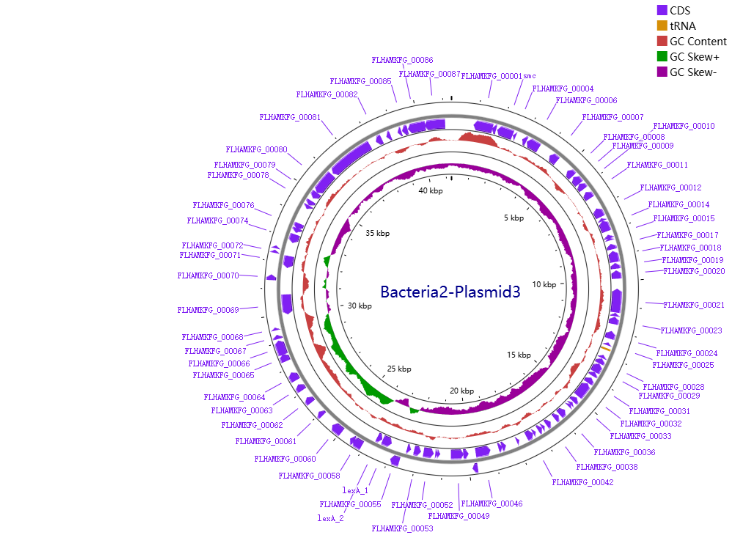
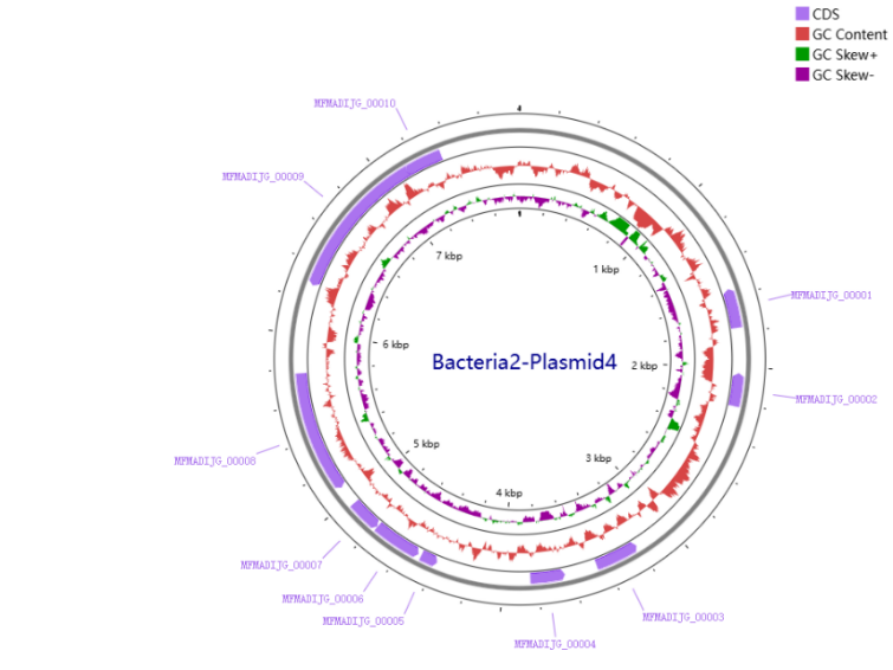
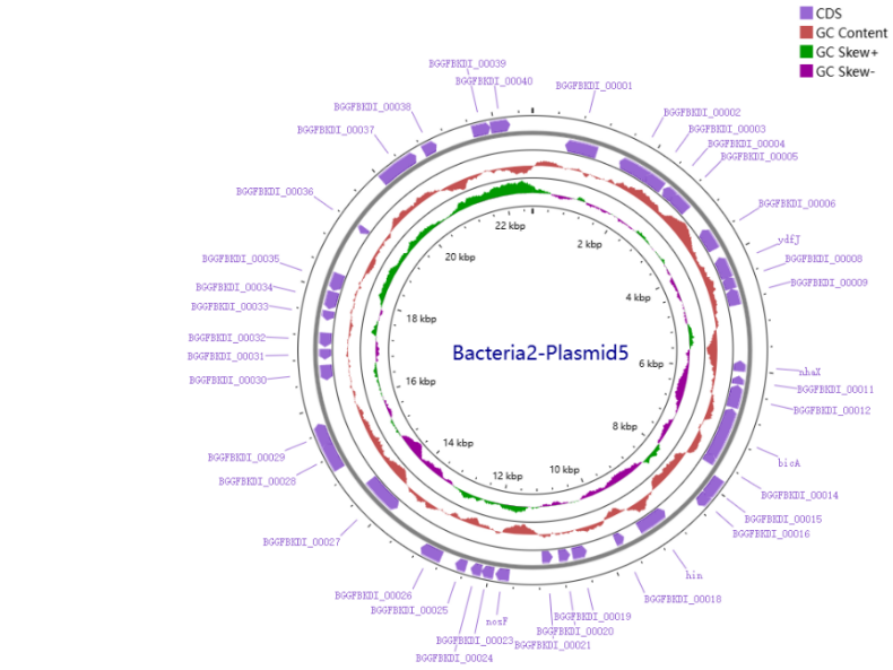
图6 bacteria2的基因组图谱
测试结论
此次测试结果显示,山东大学微生物改造技术全国重点实验室能够充分利用CycloneSEQ纳米孔测序平台的优势,完成细菌基因组组装。
测评涉及产品信息


 基因测序仪业务 SEQ ALL
基因测序仪业务 SEQ ALL











 核心技术
核心技术 热门应用
热门应用 组学研究
组学研究 文档下载
文档下载 已发表文章
已发表文章 用户成功案例
用户成功案例 在线数据集
在线数据集 多功能中心
多功能中心 客户中心
客户中心 在线支持
在线支持 关于智造
关于智造 专题推荐
专题推荐



















